Perché non è facile avere nuovi farmaci per il tumore del pancreas
4 Giugno 2017 • Redazione
Viviamo in un’epoca in cui siamo costantemente bombardati da notizie eclatanti su nuovi farmaci che offrono cure per vari tumori. Fatte salvo poche eccezioni e per pochi tumori queste notizie sono esagerazioni da parte di media e di giornalisti che attraverso titoli roboanti cercano di catturare l’attenzione del lettore. Purtroppo a volte quel lettore è un paziente o il familiare di un paziente che viene indotto a credere nella disponibilità di una cura quando, nelle migliore delle ipotesi, si sta parlando di studi di laboratorio i cui risultati verosimilmente non arriveranno mai sul banco di una farmacia. Una delle ragioni per cui ci sono poche cure disponibili per molti tumori è l’incredibile complessità del lavoro richiesto per lo di sviluppo un nuovo farmaco.
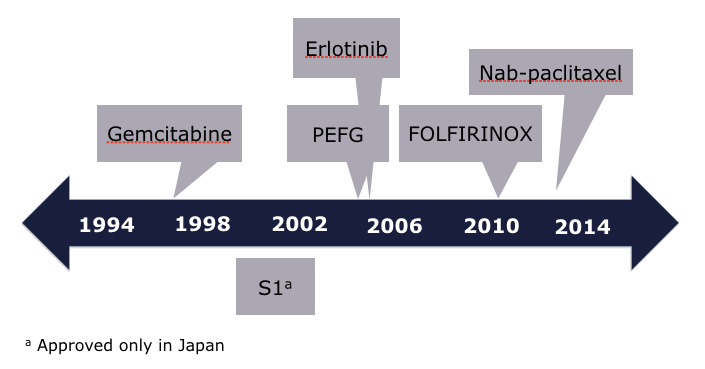
I pazienti di tumore del pancreas vivono questo problema sulla loro pelle perché hanno ben chiaro il ristretto numero di opzioni di farmaci attualmente disponibili e la lentezza con cui ne arrivano nuovi come mostrato in figura.
Nel video sottostante Riccardo Sabatini, fisico quantistico e imprenditore, illustra la complessità e la difficoltà nello sviluppo di un nuovo farmaco e indica alcune promettenti opzioni che le tecnologie digitali stanno aprendo nel mondo della ricerca dei farmaci. Il video è in inglese, il successivo testo descrive in italiano il senso della presentazione.
L’approccio corrente allo sviluppo di nuovi farmaci
Alcuni dati possono aiutarci a comprendere le dimensioni del problema. Bisogna essere consapevoli che dal punto di vista economico a fronte di 1 Miliardo di $ di investimento nello sviluppo di un nuovo farmaco nel:
- 1980 si riusciva a immettere sul mercato 10 nuovi farmaci
- 2000 si riusciva ammettere sul mercato 1 nuovo farmaco
- 2010 si riusciva ammettere sul mercato 0,5 farmaci
- 2015 si riusciva ammettere sul mercato 0,3 farmaci
In altre parole oggi per avere un nuovo farmaco bisogna investire 3.5 Miliardi $ a causa del numero di fallimenti o perché la molecola non produce l’effetto desiderato o perché alcune volte gli effetti collaterali sono talmente tossici da superare i benefici.
Questo fenomeno è noto con il nome di Eroom’s Law, http://read.bi/1Xzvhd2, ed è nato dall’osservazione statistica che il processo di scoperta e lancio sul mercato di nuovi farmaci a partire dal 1980 è via via diventato più lungo. Il nome Eroom deriva dalla lettura al contrario della parola Moore, cognome dello scienziato americano che negli anni ’80 aveva previsto che la potenza di calcolo dei computer sarebbe raddoppiata ogni 18 mesi, Moore’s Law per l’appunto. Quindi mentre nel campo digitale la potenza di calcolo si raddoppia ogni 18 mesi, nel settore della ricerca farmacologica assistiamo ad un fenomeno con tendenza contraria per l’introduzione di nuovi farmaci.
Perché accade questo? Succede per vari fattori ed anche perché il processo di sviluppo di un nuovo farmaco utilizzato attualmente è piuttosto semplicistico. Infatti per verificare l’efficacia di una nuova molecola o di una combinazione di molecole approvate, le cellule della patologia che si vuole curare vengono bombardate dalla nuova molecola per verificare i risultati prima in laboratorio, per poi passare alla sperimentazione su cavie e per infine arrivare alle sperimentazioni cliniche. Da dove vengono le molecole del nuovo farmaco? Dall’elenco delle molecole approvate e disponibili il quale non è cosi grande come potremmo pensare, è pari solo a 1.760 dopo un numero di sperimentazioni eseguite superiore alle 10.000. Numero non banale se si pensa che una sperimentazione può arrivare a costare centinaia di milioni euro. Perché questo tasso di fallimento? Perché l’attuale processo di sviluppo dei farmaci è fondamentalmente un esercizio di forza bruta di prova ed errore, hit-and-miss, per verificare la reazione della molecola nuova sulle proteine che si vogliono colpire. Non è casuale che in inglese questo processo abbia il nome library screening o drug screening. Il tutto non sempre supportato da una conoscenza soddisfacente della biologia umana. Un esempio rilevante in questo contesto? La limitata conoscenza della biologia del tumore del pancreas!
I nuovi approcci nello sviluppo dei nuovi farmaci
Negli ultimi anni le tecnologie digitali e gli approcci interdisciplinari tipici del mondo del business digitale cominciano ad avere un impatto sempre più rilevante nel mondo della medicina, https://codiceviola.org/nuovi-orizzonti/
La scoperta del DNA e la sua comprensione ha di fatto abilitato l’utilizzo intensivo delle tecnologie digitali in maniera massiccia in campo medico. Infatti il genoma umano, il nostro codice vivente, è costituito da 3 Miliardi di lettere la cui sequenziazione e lettura efficiente è resa possibile solo attraverso programmi software eseguita su calcolatori molto potenti. Infatti poiché il nostro DNA si basa su un alfabeto di 4 lettere e si presta benissimo ad essere elaborato da programmi software la cui unica e principale caratteristica è quella di elaborare codici. Nessuna magia!
Per farsi un’idea di che cosa sia il genoma si può vedere la stampa del genoma umano di una singola persona nell’immagine sottostante: 265.000 pagine!
E ognuna di queste 2650.000 pagine è una sequenza di lettere variamente combinate derivanti da un alfabeto di base fatto delle sole 4 lettere, A C G T, dove:
- A sta per adenina
- C sta per citosina
- G sta per guamina
- T sta per timina
- Stampa parziale delle sequenza del genoma umano
Per un grande ironia del caso questi potenti calcolatori non sono degli armadi enormi con tante lucine lampeggianti del nostro immaginario ma sono i processori che si trovano nelle console dei videogiochi dei ragazzini: le GPU, Graphical Processing Unit. Si! dovremo dire grazie alla comunità dei videogiochi, frivola agli occhi della comunità medica, per alcuni potenziali prossimi progressi della ricerca scientifica in campo medico.
L’1% di questi codici del genoma umano, circa 3 miliardi di lettere, sono responsabili della codifica e quindi della creazione, espressione genica, di molecole complesse che conosciamo come proteine.
Nel nostro corpo abbiamo circa 20.000 differenti proteine.
Ad esempio la proteina Esochinasi 1 attiva la glicolisi, il fondamentale processo metabolico che dagli zuccheri produce energia consumabile dal nostro corpo. Come funziona questo processo? All’interno di una proteina ci sono dei binding pocket, aree delle cellule che attraggono altre molecole, che sono in grado poi di attivare processi di rottura o di legame con altre molecole. Nel caso del processo metabolico della glicolosi la proteina Esochinesi 1 avvia il processo di scomposizione del glucosio in una catena di reazioni chimiche mostrate in figura, con i prodotti intermedi cerchiati in verde, altre proteine, che arriva a produrre la molecola che fornisce energia al nostro corpo.
Nel caso delle patologie tumorali vorremmo che le molecole dei farmaci attivassero in maniera mirata il processo di distruzione delle molecole, proteine, che concorrono allo sviluppo della malattia. Avendo ripetuto più volte la parole proteine non deve sorprendere l’attuale attenzione della ricerca nei confronti della disciplina della proteomica, proteine espresse dal genoma. L’interazione tra le varie molecole delle proteine rappresenta uno dei temi più importanti e difficili che i ricercatori devono affrontare nello sviluppo di nuovi farmaci.
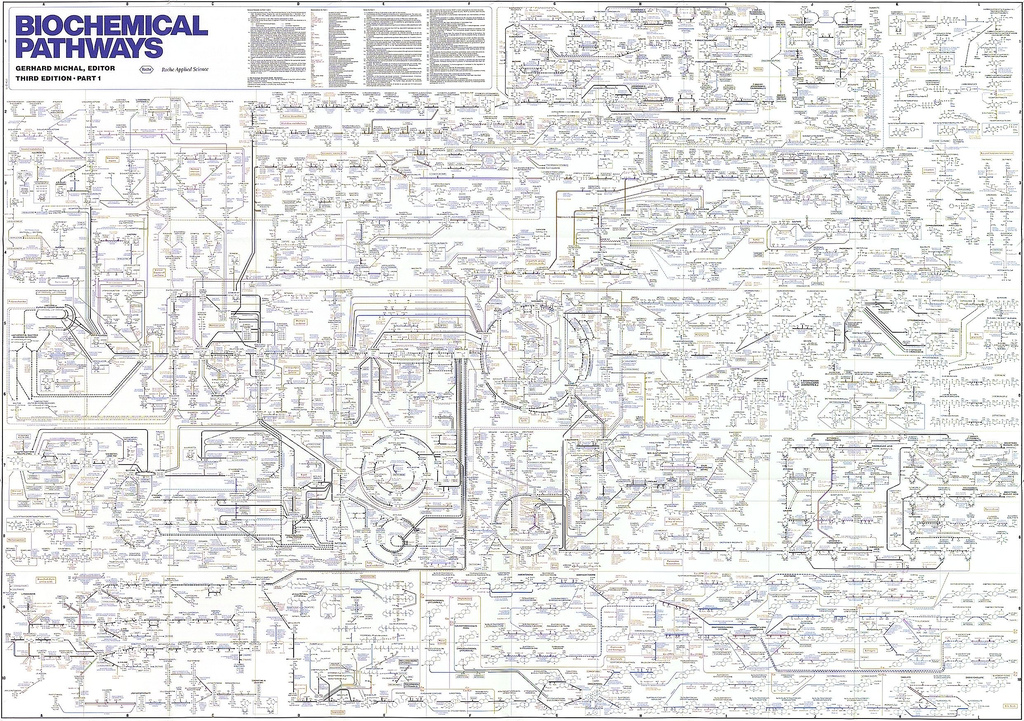
Quella sotto è la mappa di tutti i processi metabolici nel corpo umano. La vita nel corpo umano è la concertazione di tutti questi processi metabolici insieme ad altri meccanismi di funzionamento. Questa mappa e i processi chimici, che peraltro possono cambiare nel tempo, basti pensare all’invecchiamento, ci aiutano a visualizzare la complessità biologica del corpo umano con cui si devono confrontare i ricercatori e ci aiutano a capire perché è così maledettamente difficile trovare delle cure. E si badi che una mappa è sempre un modello approssimato della realtà!
Un farmaco è quindi una molecola che deve andare a colpire uno specifico punto di questa incredibile mappa, un nodo di questa rete complessa di interazioni, per innescare un reazione: attivare o disattivare una funzione, ad esempio bloccare una funzione metabolica o farla ripartire. Quindi si può intuire la gigantesca difficoltà nello sviluppo di un nuovo farmaco perché si deve arrivare in uno specifico punto della mappa attraversando una serie di percorsi metabolici senza innescare degli effetti secondari indesiderati in altri punti della mappa. Come fare? Pensando alle 3 miliardi di lettere del genoma e alla complessità della mappa dei processi metabolici si intuisce che il numero di di possibili interazioni o ipotesi da verificare è sbalorditivamente alto. Ed è qui che entrano in gioco le tecnologie digitali. Dal punto di vista dell’hardware abbiamo a disposizione non solo calcolatori più potenti ma anche pensati per fare tanti calcoli in parallelo, le GPU. Dal punto di vista del software a partire dal 2012 si sono affermati dei metodi di calcolo, algoritmi, che sono in grado in grado di apprendere e classificare dati e comportamenti partendo da vaste mole di dati. Queste tecniche sono note sotto il nome di machine learning, http://bit.ly/2s6BiEp.
Il nuovo approccio prevede di prendere in considerazione la molecola del nuovo farmaco, la sequenziazione genomica di un individuo e la mappa dei processi metabolici per verificare dove questa molecola interagisce nel nostro corpo. Queste interazioni vengono poi classificate in 3 tipi:
- molecole che colpiscono la proteina target, l’obiettivo
- molecole che colpiscono proteine note ma che non sono il target
- molecole che colpiscono proteine di cui non conosciamo tutto o proteine che hanno effetti collaterali disastrosi
L’obiettivo che si propone il progetto è l’analisi tutti i farmaci disponibili, tutti quelli che hanno fallito e altri prodotti naturali e verificare quali molecole attivano nel genoma umano, in altre parole si vuole verificare come i farmaci interagiscono su scala genomica con il nostro corpo. In questo modo si creerebbe una piattaforma su cui testare più velocemente delle ipotesi per un nuovo farmaco.
Conclusioni
Un primo aspetto importante: nella ricerca di nuovi farmaci contro i tumori la conoscenza biologica del comportamento proteine è fondamentale. L’informazione generale tende sempre a mettere in evidenza la sola importanza del DNA e del genoma trascurando il fatto che i farmaci sono delle molecole che interagiscono con delle proteine che per loro natura sono una espressione genica.
Se si riuscirà a comprendere meglio come le molecole dei farmaci interagiscono con le proteine del nostro corpo utilizzando tecniche di machine-learning potremmo idealmente essere in grado di prevedere meglio la probabilità di successo di un nuovo farmaco. Questa è una delle scommesse del futuro e rappresenta un grande cambiamento rispetto all’approccio classico dello sviluppo dei farmaci. Bisogna evitare facili illusioni: in questo campo misureremo i risultati reali per i pazienti non nell’ordine dei mesi o dell’anno ma nell’ordine degli anni.
E in questo contesto esiste qualche prospettiva nella ricerca sul tumore del pancreas?
A Boston l’azienda di biotecnologie Berg sta utilizzando tecniche per lo sviluppo di un farmaco simili a quelle descritte nel video per verificare l’impatto di una molecola sul tumore del pancreas, http://www.wired.co.uk/article/ai-cancer-drugs-berg-pharma-startup.
Il Memorial Sloan Kettering Center, MSK, uno dei maggiori centri mondiali nella ricerca del tumore del pancreas, ha recentissimamente deciso di utilizzare la piattaforma Intellispace Genomics della Philips per le ricerche sulla diagnosi e le terapie per il tumore del pancreas: http://bit.ly/2qKy9Ky.
Un’osservazione finale: la complessità di patologie come il tumore del pancreas confermano la necessità di un approccio interdisciplinare tra medici, biologi, biotecnologi e bioinformatici. I pazienti tendono a idealizzare alcune figure, in particolare il chirurgo, ma in queste patologie non esiste il deus ex machina che da solo troverà la cura del tumore del pancreas.
Condividi questo articolo

Perché non è facile avere nuovi farmaci per il tumore del pancreas
4 Giugno 2017 • Redazione
Viviamo in un’epoca in cui siamo costantemente bombardati da notizie eclatanti su nuovi farmaci che offrono cure per vari tumori. Fatte salvo poche eccezioni e per pochi tumori queste notizie sono esagerazioni da parte di media e di giornalisti che attraverso titoli roboanti cercano di catturare l’attenzione del lettore. Purtroppo a volte quel lettore è un paziente o il familiare di un paziente che viene indotto a credere nella disponibilità di una cura quando, nelle migliore delle ipotesi, si sta parlando di studi di laboratorio i cui risultati verosimilmente non arriveranno mai sul banco di una farmacia. Una delle ragioni per cui ci sono poche cure disponibili per molti tumori è l’incredibile complessità del lavoro richiesto per lo di sviluppo un nuovo farmaco.
I pazienti di tumore del pancreas vivono questo problema sulla loro pelle perché hanno ben chiaro il ristretto numero di opzioni di farmaci attualmente disponibili e la lentezza con cui ne arrivano nuovi come mostrato in figura.
Nel video sottostante Riccardo Sabatini, fisico quantistico e imprenditore, illustra la complessità e la difficoltà nello sviluppo di un nuovo farmaco e indica alcune promettenti opzioni che le tecnologie digitali stanno aprendo nel mondo della ricerca dei farmaci. Il video è in inglese, il successivo testo descrive in italiano il senso della presentazione.
L’approccio corrente allo sviluppo di nuovi farmaci
Alcuni dati possono aiutarci a comprendere le dimensioni del problema. Bisogna essere consapevoli che dal punto di vista economico a fronte di 1 Miliardo di $ di investimento nello sviluppo di un nuovo farmaco nel:
- 1980 si riusciva a immettere sul mercato 10 nuovi farmaci
- 2000 si riusciva ammettere sul mercato 1 nuovo farmaco
- 2010 si riusciva ammettere sul mercato 0,5 farmaci
- 2015 si riusciva ammettere sul mercato 0,3 farmaci
In altre parole oggi per avere un nuovo farmaco bisogna investire 3.5 Miliardi $ a causa del numero di fallimenti o perché la molecola non produce l’effetto desiderato o perché alcune volte gli effetti collaterali sono talmente tossici da superare i benefici.
Questo fenomeno è noto con il nome di Eroom’s Law, http://read.bi/1Xzvhd2, ed è nato dall’osservazione statistica che il processo di scoperta e lancio sul mercato di nuovi farmaci a partire dal 1980 è via via diventato più lungo. Il nome Eroom deriva dalla lettura al contrario della parola Moore, cognome dello scienziato americano che negli anni ’80 aveva previsto che la potenza di calcolo dei computer sarebbe raddoppiata ogni 18 mesi, Moore’s Law per l’appunto. Quindi mentre nel campo digitale la potenza di calcolo si raddoppia ogni 18 mesi, nel settore della ricerca farmacologica assistiamo ad un fenomeno con tendenza contraria per l’introduzione di nuovi farmaci.
Perché accade questo? Succede per vari fattori ed anche perché il processo di sviluppo di un nuovo farmaco utilizzato attualmente è piuttosto semplicistico. Infatti per verificare l’efficacia di una nuova molecola o di una combinazione di molecole approvate, le cellule della patologia che si vuole curare vengono bombardate dalla nuova molecola per verificare i risultati prima in laboratorio, per poi passare alla sperimentazione su cavie e per infine arrivare alle sperimentazioni cliniche. Da dove vengono le molecole del nuovo farmaco? Dall’elenco delle molecole approvate e disponibili il quale non è cosi grande come potremmo pensare, è pari solo a 1.760 dopo un numero di sperimentazioni eseguite superiore alle 10.000. Numero non banale se si pensa che una sperimentazione può arrivare a costare centinaia di milioni euro. Perché questo tasso di fallimento? Perché l’attuale processo di sviluppo dei farmaci è fondamentalmente un esercizio di forza bruta di prova ed errore, hit-and-miss, per verificare la reazione della molecola nuova sulle proteine che si vogliono colpire. Non è casuale che in inglese questo processo abbia il nome library screening o drug screening. Il tutto non sempre supportato da una conoscenza soddisfacente della biologia umana. Un esempio rilevante in questo contesto? La limitata conoscenza della biologia del tumore del pancreas!
I nuovi approcci nello sviluppo dei nuovi farmaci
Negli ultimi anni le tecnologie digitali e gli approcci interdisciplinari tipici del mondo del business digitale cominciano ad avere un impatto sempre più rilevante nel mondo della medicina, https://codiceviola.org/nuovi-orizzonti/
La scoperta del DNA e la sua comprensione ha di fatto abilitato l’utilizzo intensivo delle tecnologie digitali in maniera massiccia in campo medico. Infatti il genoma umano, il nostro codice vivente, è costituito da 3 Miliardi di lettere la cui sequenziazione e lettura efficiente è resa possibile solo attraverso programmi software eseguita su calcolatori molto potenti. Infatti poiché il nostro DNA si basa su un alfabeto di 4 lettere e si presta benissimo ad essere elaborato da programmi software la cui unica e principale caratteristica è quella di elaborare codici. Nessuna magia!
Per farsi un’idea di che cosa sia il genoma si può vedere la stampa del genoma umano di una singola persona nell’immagine sottostante: 265.000 pagine!
E ognuna di queste 2650.000 pagine è una sequenza di lettere variamente combinate derivanti da un alfabeto di base fatto delle sole 4 lettere, A C G T, dove:
- A sta per adenina
- C sta per citosina
- G sta per guamina
- T sta per timina
- Stampa parziale delle sequenza del genoma umano
Per un grande ironia del caso questi potenti calcolatori non sono degli armadi enormi con tante lucine lampeggianti del nostro immaginario ma sono i processori che si trovano nelle console dei videogiochi dei ragazzini: le GPU, Graphical Processing Unit. Si! dovremo dire grazie alla comunità dei videogiochi, frivola agli occhi della comunità medica, per alcuni potenziali prossimi progressi della ricerca scientifica in campo medico.
L’1% di questi codici del genoma umano, circa 3 miliardi di lettere, sono responsabili della codifica e quindi della creazione, espressione genica, di molecole complesse che conosciamo come proteine.
Nel nostro corpo abbiamo circa 20.000 differenti proteine.
Ad esempio la proteina Esochinasi 1 attiva la glicolisi, il fondamentale processo metabolico che dagli zuccheri produce energia consumabile dal nostro corpo. Come funziona questo processo? All’interno di una proteina ci sono dei binding pocket, aree delle cellule che attraggono altre molecole, che sono in grado poi di attivare processi di rottura o di legame con altre molecole. Nel caso del processo metabolico della glicolosi la proteina Esochinesi 1 avvia il processo di scomposizione del glucosio in una catena di reazioni chimiche mostrate in figura, con i prodotti intermedi cerchiati in verde, altre proteine, che arriva a produrre la molecola che fornisce energia al nostro corpo.
Nel caso delle patologie tumorali vorremmo che le molecole dei farmaci attivassero in maniera mirata il processo di distruzione delle molecole, proteine, che concorrono allo sviluppo della malattia. Avendo ripetuto più volte la parole proteine non deve sorprendere l’attuale attenzione della ricerca nei confronti della disciplina della proteomica, proteine espresse dal genoma. L’interazione tra le varie molecole delle proteine rappresenta uno dei temi più importanti e difficili che i ricercatori devono affrontare nello sviluppo di nuovi farmaci.
Quella sotto è la mappa di tutti i processi metabolici nel corpo umano. La vita nel corpo umano è la concertazione di tutti questi processi metabolici insieme ad altri meccanismi di funzionamento. Questa mappa e i processi chimici, che peraltro possono cambiare nel tempo, basti pensare all’invecchiamento, ci aiutano a visualizzare la complessità biologica del corpo umano con cui si devono confrontare i ricercatori e ci aiutano a capire perché è così maledettamente difficile trovare delle cure. E si badi che una mappa è sempre un modello approssimato della realtà!
Un farmaco è quindi una molecola che deve andare a colpire uno specifico punto di questa incredibile mappa, un nodo di questa rete complessa di interazioni, per innescare un reazione: attivare o disattivare una funzione, ad esempio bloccare una funzione metabolica o farla ripartire. Quindi si può intuire la gigantesca difficoltà nello sviluppo di un nuovo farmaco perché si deve arrivare in uno specifico punto della mappa attraversando una serie di percorsi metabolici senza innescare degli effetti secondari indesiderati in altri punti della mappa. Come fare? Pensando alle 3 miliardi di lettere del genoma e alla complessità della mappa dei processi metabolici si intuisce che il numero di di possibili interazioni o ipotesi da verificare è sbalorditivamente alto. Ed è qui che entrano in gioco le tecnologie digitali. Dal punto di vista dell’hardware abbiamo a disposizione non solo calcolatori più potenti ma anche pensati per fare tanti calcoli in parallelo, le GPU. Dal punto di vista del software a partire dal 2012 si sono affermati dei metodi di calcolo, algoritmi, che sono in grado in grado di apprendere e classificare dati e comportamenti partendo da vaste mole di dati. Queste tecniche sono note sotto il nome di machine learning, http://bit.ly/2s6BiEp.
Il nuovo approccio prevede di prendere in considerazione la molecola del nuovo farmaco, la sequenziazione genomica di un individuo e la mappa dei processi metabolici per verificare dove questa molecola interagisce nel nostro corpo. Queste interazioni vengono poi classificate in 3 tipi:
- molecole che colpiscono la proteina target, l’obiettivo
- molecole che colpiscono proteine note ma che non sono il target
- molecole che colpiscono proteine di cui non conosciamo tutto o proteine che hanno effetti collaterali disastrosi
L’obiettivo che si propone il progetto è l’analisi tutti i farmaci disponibili, tutti quelli che hanno fallito e altri prodotti naturali e verificare quali molecole attivano nel genoma umano, in altre parole si vuole verificare come i farmaci interagiscono su scala genomica con il nostro corpo. In questo modo si creerebbe una piattaforma su cui testare più velocemente delle ipotesi per un nuovo farmaco.
Conclusioni
Un primo aspetto importante: nella ricerca di nuovi farmaci contro i tumori la conoscenza biologica del comportamento proteine è fondamentale. L’informazione generale tende sempre a mettere in evidenza la sola importanza del DNA e del genoma trascurando il fatto che i farmaci sono delle molecole che interagiscono con delle proteine che per loro natura sono una espressione genica.
Se si riuscirà a comprendere meglio come le molecole dei farmaci interagiscono con le proteine del nostro corpo utilizzando tecniche di machine-learning potremmo idealmente essere in grado di prevedere meglio la probabilità di successo di un nuovo farmaco. Questa è una delle scommesse del futuro e rappresenta un grande cambiamento rispetto all’approccio classico dello sviluppo dei farmaci. Bisogna evitare facili illusioni: in questo campo misureremo i risultati reali per i pazienti non nell’ordine dei mesi o dell’anno ma nell’ordine degli anni.
E in questo contesto esiste qualche prospettiva nella ricerca sul tumore del pancreas?
A Boston l’azienda di biotecnologie Berg sta utilizzando tecniche per lo sviluppo di un farmaco simili a quelle descritte nel video per verificare l’impatto di una molecola sul tumore del pancreas, http://www.wired.co.uk/article/ai-cancer-drugs-berg-pharma-startup.
Il Memorial Sloan Kettering Center, MSK, uno dei maggiori centri mondiali nella ricerca del tumore del pancreas, ha recentissimamente deciso di utilizzare la piattaforma Intellispace Genomics della Philips per le ricerche sulla diagnosi e le terapie per il tumore del pancreas: http://bit.ly/2qKy9Ky.
Un’osservazione finale: la complessità di patologie come il tumore del pancreas confermano la necessità di un approccio interdisciplinare tra medici, biologi, biotecnologi e bioinformatici. I pazienti tendono a idealizzare alcune figure, in particolare il chirurgo, ma in queste patologie non esiste il deus ex machina che da solo troverà la cura del tumore del pancreas.
Condividi questo articolo

Perché non è facile avere nuovi farmaci per il tumore del pancreas
4 Giugno 2017 • Redazione
Viviamo in un’epoca in cui siamo costantemente bombardati da notizie eclatanti su nuovi farmaci che offrono cure per vari tumori. Fatte salvo poche eccezioni e per pochi tumori queste notizie sono esagerazioni da parte di media e di giornalisti che attraverso titoli roboanti cercano di catturare l’attenzione del lettore. Purtroppo a volte quel lettore è un paziente o il familiare di un paziente che viene indotto a credere nella disponibilità di una cura quando, nelle migliore delle ipotesi, si sta parlando di studi di laboratorio i cui risultati verosimilmente non arriveranno mai sul banco di una farmacia. Una delle ragioni per cui ci sono poche cure disponibili per molti tumori è l’incredibile complessità del lavoro richiesto per lo di sviluppo un nuovo farmaco.
I pazienti di tumore del pancreas vivono questo problema sulla loro pelle perché hanno ben chiaro il ristretto numero di opzioni di farmaci attualmente disponibili e la lentezza con cui ne arrivano nuovi come mostrato in figura.
Nel video sottostante Riccardo Sabatini, fisico quantistico e imprenditore, illustra la complessità e la difficoltà nello sviluppo di un nuovo farmaco e indica alcune promettenti opzioni che le tecnologie digitali stanno aprendo nel mondo della ricerca dei farmaci. Il video è in inglese, il successivo testo descrive in italiano il senso della presentazione.
L’approccio corrente allo sviluppo di nuovi farmaci
Alcuni dati possono aiutarci a comprendere le dimensioni del problema. Bisogna essere consapevoli che dal punto di vista economico a fronte di 1 Miliardo di $ di investimento nello sviluppo di un nuovo farmaco nel:
- 1980 si riusciva a immettere sul mercato 10 nuovi farmaci
- 2000 si riusciva ammettere sul mercato 1 nuovo farmaco
- 2010 si riusciva ammettere sul mercato 0,5 farmaci
- 2015 si riusciva ammettere sul mercato 0,3 farmaci
In altre parole oggi per avere un nuovo farmaco bisogna investire 3.5 Miliardi $ a causa del numero di fallimenti o perché la molecola non produce l’effetto desiderato o perché alcune volte gli effetti collaterali sono talmente tossici da superare i benefici.
Questo fenomeno è noto con il nome di Eroom’s Law, http://read.bi/1Xzvhd2, ed è nato dall’osservazione statistica che il processo di scoperta e lancio sul mercato di nuovi farmaci a partire dal 1980 è via via diventato più lungo. Il nome Eroom deriva dalla lettura al contrario della parola Moore, cognome dello scienziato americano che negli anni ’80 aveva previsto che la potenza di calcolo dei computer sarebbe raddoppiata ogni 18 mesi, Moore’s Law per l’appunto. Quindi mentre nel campo digitale la potenza di calcolo si raddoppia ogni 18 mesi, nel settore della ricerca farmacologica assistiamo ad un fenomeno con tendenza contraria per l’introduzione di nuovi farmaci.
Perché accade questo? Succede per vari fattori ed anche perché il processo di sviluppo di un nuovo farmaco utilizzato attualmente è piuttosto semplicistico. Infatti per verificare l’efficacia di una nuova molecola o di una combinazione di molecole approvate, le cellule della patologia che si vuole curare vengono bombardate dalla nuova molecola per verificare i risultati prima in laboratorio, per poi passare alla sperimentazione su cavie e per infine arrivare alle sperimentazioni cliniche. Da dove vengono le molecole del nuovo farmaco? Dall’elenco delle molecole approvate e disponibili il quale non è cosi grande come potremmo pensare, è pari solo a 1.760 dopo un numero di sperimentazioni eseguite superiore alle 10.000. Numero non banale se si pensa che una sperimentazione può arrivare a costare centinaia di milioni euro. Perché questo tasso di fallimento? Perché l’attuale processo di sviluppo dei farmaci è fondamentalmente un esercizio di forza bruta di prova ed errore, hit-and-miss, per verificare la reazione della molecola nuova sulle proteine che si vogliono colpire. Non è casuale che in inglese questo processo abbia il nome library screening o drug screening. Il tutto non sempre supportato da una conoscenza soddisfacente della biologia umana. Un esempio rilevante in questo contesto? La limitata conoscenza della biologia del tumore del pancreas!
I nuovi approcci nello sviluppo dei nuovi farmaci
Negli ultimi anni le tecnologie digitali e gli approcci interdisciplinari tipici del mondo del business digitale cominciano ad avere un impatto sempre più rilevante nel mondo della medicina, https://codiceviola.org/nuovi-orizzonti/
La scoperta del DNA e la sua comprensione ha di fatto abilitato l’utilizzo intensivo delle tecnologie digitali in maniera massiccia in campo medico. Infatti il genoma umano, il nostro codice vivente, è costituito da 3 Miliardi di lettere la cui sequenziazione e lettura efficiente è resa possibile solo attraverso programmi software eseguita su calcolatori molto potenti. Infatti poiché il nostro DNA si basa su un alfabeto di 4 lettere e si presta benissimo ad essere elaborato da programmi software la cui unica e principale caratteristica è quella di elaborare codici. Nessuna magia!
Per farsi un’idea di che cosa sia il genoma si può vedere la stampa del genoma umano di una singola persona nell’immagine sottostante: 265.000 pagine!
E ognuna di queste 2650.000 pagine è una sequenza di lettere variamente combinate derivanti da un alfabeto di base fatto delle sole 4 lettere, A C G T, dove:
- A sta per adenina
- C sta per citosina
- G sta per guamina
- T sta per timina
- Stampa parziale delle sequenza del genoma umano
Per un grande ironia del caso questi potenti calcolatori non sono degli armadi enormi con tante lucine lampeggianti del nostro immaginario ma sono i processori che si trovano nelle console dei videogiochi dei ragazzini: le GPU, Graphical Processing Unit. Si! dovremo dire grazie alla comunità dei videogiochi, frivola agli occhi della comunità medica, per alcuni potenziali prossimi progressi della ricerca scientifica in campo medico.
L’1% di questi codici del genoma umano, circa 3 miliardi di lettere, sono responsabili della codifica e quindi della creazione, espressione genica, di molecole complesse che conosciamo come proteine.
Nel nostro corpo abbiamo circa 20.000 differenti proteine.
Ad esempio la proteina Esochinasi 1 attiva la glicolisi, il fondamentale processo metabolico che dagli zuccheri produce energia consumabile dal nostro corpo. Come funziona questo processo? All’interno di una proteina ci sono dei binding pocket, aree delle cellule che attraggono altre molecole, che sono in grado poi di attivare processi di rottura o di legame con altre molecole. Nel caso del processo metabolico della glicolosi la proteina Esochinesi 1 avvia il processo di scomposizione del glucosio in una catena di reazioni chimiche mostrate in figura, con i prodotti intermedi cerchiati in verde, altre proteine, che arriva a produrre la molecola che fornisce energia al nostro corpo.
Nel caso delle patologie tumorali vorremmo che le molecole dei farmaci attivassero in maniera mirata il processo di distruzione delle molecole, proteine, che concorrono allo sviluppo della malattia. Avendo ripetuto più volte la parole proteine non deve sorprendere l’attuale attenzione della ricerca nei confronti della disciplina della proteomica, proteine espresse dal genoma. L’interazione tra le varie molecole delle proteine rappresenta uno dei temi più importanti e difficili che i ricercatori devono affrontare nello sviluppo di nuovi farmaci.
Quella sotto è la mappa di tutti i processi metabolici nel corpo umano. La vita nel corpo umano è la concertazione di tutti questi processi metabolici insieme ad altri meccanismi di funzionamento. Questa mappa e i processi chimici, che peraltro possono cambiare nel tempo, basti pensare all’invecchiamento, ci aiutano a visualizzare la complessità biologica del corpo umano con cui si devono confrontare i ricercatori e ci aiutano a capire perché è così maledettamente difficile trovare delle cure. E si badi che una mappa è sempre un modello approssimato della realtà!
Un farmaco è quindi una molecola che deve andare a colpire uno specifico punto di questa incredibile mappa, un nodo di questa rete complessa di interazioni, per innescare un reazione: attivare o disattivare una funzione, ad esempio bloccare una funzione metabolica o farla ripartire. Quindi si può intuire la gigantesca difficoltà nello sviluppo di un nuovo farmaco perché si deve arrivare in uno specifico punto della mappa attraversando una serie di percorsi metabolici senza innescare degli effetti secondari indesiderati in altri punti della mappa. Come fare? Pensando alle 3 miliardi di lettere del genoma e alla complessità della mappa dei processi metabolici si intuisce che il numero di di possibili interazioni o ipotesi da verificare è sbalorditivamente alto. Ed è qui che entrano in gioco le tecnologie digitali. Dal punto di vista dell’hardware abbiamo a disposizione non solo calcolatori più potenti ma anche pensati per fare tanti calcoli in parallelo, le GPU. Dal punto di vista del software a partire dal 2012 si sono affermati dei metodi di calcolo, algoritmi, che sono in grado in grado di apprendere e classificare dati e comportamenti partendo da vaste mole di dati. Queste tecniche sono note sotto il nome di machine learning, http://bit.ly/2s6BiEp.
Il nuovo approccio prevede di prendere in considerazione la molecola del nuovo farmaco, la sequenziazione genomica di un individuo e la mappa dei processi metabolici per verificare dove questa molecola interagisce nel nostro corpo. Queste interazioni vengono poi classificate in 3 tipi:
- molecole che colpiscono la proteina target, l’obiettivo
- molecole che colpiscono proteine note ma che non sono il target
- molecole che colpiscono proteine di cui non conosciamo tutto o proteine che hanno effetti collaterali disastrosi
L’obiettivo che si propone il progetto è l’analisi tutti i farmaci disponibili, tutti quelli che hanno fallito e altri prodotti naturali e verificare quali molecole attivano nel genoma umano, in altre parole si vuole verificare come i farmaci interagiscono su scala genomica con il nostro corpo. In questo modo si creerebbe una piattaforma su cui testare più velocemente delle ipotesi per un nuovo farmaco.
Conclusioni
Un primo aspetto importante: nella ricerca di nuovi farmaci contro i tumori la conoscenza biologica del comportamento proteine è fondamentale. L’informazione generale tende sempre a mettere in evidenza la sola importanza del DNA e del genoma trascurando il fatto che i farmaci sono delle molecole che interagiscono con delle proteine che per loro natura sono una espressione genica.
Se si riuscirà a comprendere meglio come le molecole dei farmaci interagiscono con le proteine del nostro corpo utilizzando tecniche di machine-learning potremmo idealmente essere in grado di prevedere meglio la probabilità di successo di un nuovo farmaco. Questa è una delle scommesse del futuro e rappresenta un grande cambiamento rispetto all’approccio classico dello sviluppo dei farmaci. Bisogna evitare facili illusioni: in questo campo misureremo i risultati reali per i pazienti non nell’ordine dei mesi o dell’anno ma nell’ordine degli anni.
E in questo contesto esiste qualche prospettiva nella ricerca sul tumore del pancreas?
A Boston l’azienda di biotecnologie Berg sta utilizzando tecniche per lo sviluppo di un farmaco simili a quelle descritte nel video per verificare l’impatto di una molecola sul tumore del pancreas, http://www.wired.co.uk/article/ai-cancer-drugs-berg-pharma-startup.
Il Memorial Sloan Kettering Center, MSK, uno dei maggiori centri mondiali nella ricerca del tumore del pancreas, ha recentissimamente deciso di utilizzare la piattaforma Intellispace Genomics della Philips per le ricerche sulla diagnosi e le terapie per il tumore del pancreas: http://bit.ly/2qKy9Ky.
Un’osservazione finale: la complessità di patologie come il tumore del pancreas confermano la necessità di un approccio interdisciplinare tra medici, biologi, biotecnologi e bioinformatici. I pazienti tendono a idealizzare alcune figure, in particolare il chirurgo, ma in queste patologie non esiste il deus ex machina che da solo troverà la cura del tumore del pancreas.